Fusie van GEDI-structuurdata met Sentinel-1 en -2 data
GEDI-datasets zijn discreet: de missie verzamelt slechts samples van het aardoppervlak in de vorm van shots. Voor gebiedsdekkende informatie, zoals kroonhoogte (rh98) of biomassa (agbd), moeten we extrapoleren. Dit kan door GEDI-gegevens te combineren met Sentinel-1 en Sentinel-2 data. In dit voorbeeld maken we een eenvoudig regressiemodel om een kroonhoogtemodel te maken. Dit proces wordt hieronder uitgelegd en geïllustreerd:
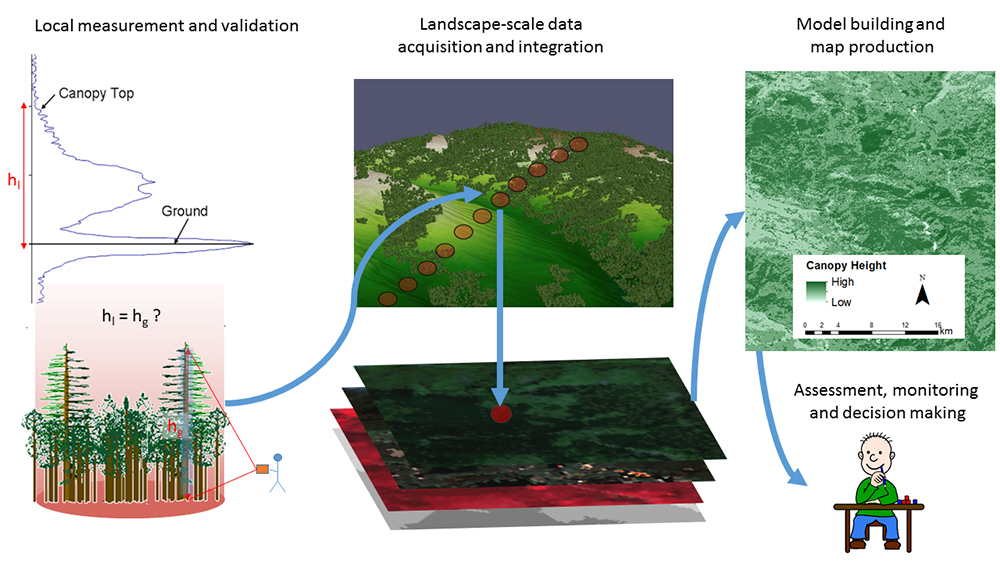
Door het fuseren van Sentinel-data met de discrete GEDI-data kunnen regionale modellen worden gemaakt.
Toevoegen van Sentinel-1 en Sentinel-2 data
De onderstaand stappenplan voegt maakt de Sentinel-1- en Sentinel-2-beelden aan voor het studiegebied (ROI). Hierbij worden de volgende stappen uitgevoerd:
- Gefilterd op het jaar 2021 en wolkbedekking van maximaal 50%
- Wolken werden verwijderd
- Voor Sentinel-1: 'speckle' filter toegepast.
- Mediaan-reducer op resterende beelden
Om de rekentijd te minimaliseren, maken we gebruik van een reeds aangemaakt Sentinel-2-beeld voor het jaar 2021. Dit beeld is al beschikbaar in de Google Earth Engine-cloud, waardoor geen extra achterliggende berekeningen nodig zijn. Dit bespaart aanzienlijk tijd en rekencapaciteit.
// Inladen van een vooraf aangemaakt Sentinel-1/2 beeld (dit kun je vervangen door je eigen beeld)
var Sentinel_2021 = ee.Image('projects/ee-mangroves-suriname/assets/Sentinel_2021_NOTEXT')
// Visualisatie van een RGB-beeld:
Map.addLayer(Sentinel_2021, {min:0, max:0.3, bands: ['B4','B3','B2']}, 'Sentinel 2021')
Stap 1 - GEDI/Sentinel-1/2 dataset opstellen
1.1 - Converteer GEDI-pixels naar een FeatureCollection
Voor modelleren van de GEDI-afgeleide structuurdata functioneren de GEDI-shots zowel als training en validatie-dataset voor het maken van een regressie. Daarom dienen we de volledige dataset op te splitsen in een training-set (voor het aanmaken en trainen van het model) en validatie-set (om de accuraatheid van het model te kwantificeren).
Omdat we een model willen trainen, moeten we GEDI-pixels (1 pixel = 1 shot) omzetten naar punten (1 feature = 1 shot). Dit kan met de .sample()-functie.
Toepassen van de .sample()-functie
Ga in de Docs na wat de .sample()-functie doeten welke parameters nodig zijn. Gebruik region = ROI, scale = 25, geometries = true).
Oplossing
// We dienen onze GEDI-pixels om te zetten naar een ```FeatureCollection```, zodat we beschikken over een 'puntenset'
//Via de .sample-functie, plaatsen we over elke pixel een punt
var GEDI_L2A = GEDI_L2A.select('rh98').median().sample({
region: ROI,
scale: 25,
geometries: true
});
// Over hoeveel GEDI-punten beschikken we?
print('Aantal GEDI-punten :', GEDI_L2A.size())
1.2 - Dataset subsetten (30% gebruiken)
Omdat de dataset erg groot kan zijn, beperken we deze voor snellere berekeningen door een willekeurige subset van 30% te nemen.
// Toevoegen van randomColumn; deze voegt een 'attribuut' toe met getal tussen 0 en 1
var GEDI_L2A = GEDI_L2A.randomColumn('random') // De kolom noemen we 'random'
// Filteren op 'random', zodat we ongeveer 30% overhouden
var GEDI_L2A = GEDI_L2A.filter(ee.Filter.lt('random',0.3))
// Grootte dataset na deze reductie
print(GEDI_L2A.size(), ' punten na reductie')
1.3 - Pixelwaarden extraheren uit de Sentinel-1/2 dataset
Extraheer de pixel-waarden uit de Sentinel-1/2 dataset d.m.v. .SampleRegions() Dit is een gelijkaardige stap als bij het aanmaken van de trainingdataset voor een classificatie.
Oplossing
// Get bandNames
var bands = Sentinel_2021.bandNames()
// Extract data
var GEDI_S2 = Sentinel_2021.select(bands).sampleRegions({
collection: GEDI_L2A,
properties: ['rh98'],
scale: 25 ,
geometries : true,
});
Dit levert een nieuwe dataset op met volgende gegevens:
print('GEDI Dataset :' , GEDI_S2.first().propertyNames())
Stap 2 - Dataset splitsen in train & validatie data
Vervolgens kunnen we de datset opsplitsen in een training- en validatieset. Dit doen we eveneens door het toevoegen van een random kolom, waarbij we de datset willekeurig splitsen in 70% traindata en 30% validatiedata.
Maak opnieuw gebruik van .randomColumn().
Oplossing
// Toevoegen van randomColumn nr 2
var GEDI_S2 = GEDI_S2.randomColumn('random2')
// Opsplitsen 30 - 70
var training = GEDI_S2.filter(ee.Filter.lt('random2',0.7))
var validation = GEDI_S2.filter(ee.Filter.gte('random2',0.7))
Stap 3 - Aanmaken van een regressiemodel
3.1 - Model trainen
Maak een Random Forest-regressiemodel aan om kroonhoogte (rh98) te voorspellen op basis van Sentinel-1/2 banden. Deze is gelijk aan de Random Forest classifier, alleen dien je aan te geven dat we in dit geval te maken hebben met een regressie. Dit doe je door de functie .setOutputMode('REGRESSION') toe te passen op het model.
Vul onderstaande code aan zodat je het regressie-model traint op basis van de training-dataset (gelijkaardig aan een classificatie):
// Build Random Forest regressor
var RF_regressor = ee.Classifier.smileRandomForest(100) // RF regressor met ntrees = 100
.setOutputMode('REGRESSION') // Classifier -> regressor voor continue data
// ... vul aan
Oplossing
// Build Random Forest regressor
var RF_regressor = ee.Classifier.smileRandomForest(100)
.setOutputMode('REGRESSION')
.train({
features: training,
classProperty: 'rh98',
inputProperties: bands
});
3.2 - Model toepassen -> regressie uitvoeren
Pas het getrainde model toe op het volledige Sentinel-1/2- beeld maak een visualisate van het resultaat:
Oplossing
// Regressie toepassen, finale band hernoemen naar 'predicted' (zie ```.classify()```)
var regression = Sentinel_2021.select(bands).classify(RF_regressor , 'predicted');
// Afbeelden van regressieraster (zelf nog visualisatieparameters toe te voegen!)
Map.addLayer(regression, {}, 'Regression');
Stap 4 - Evaluatie van het model - de R²-waarde
Tot slot kunnen we het model evalueren aan de hand van de onafhankelijke validatiedataset (validation) die we eerder hebben aangemaakt. Voor deze dataset laten we het model voorspellingen maken en vergelijken we deze met de werkelijke waarden.
De R²-waarde (coefficient of determination) is een statistische maatstaf die wordt gebruikt om de mate van variabiliteit in de afhankelijke variabele (in dit geval rh98) te verklaren door de onafhankelijke variabelen (de Sentinel-1/2 banden) in het regressiemodel.
In eenvoudige termen geeft de R²-waarde aan hoe goed de voorspelde waarden van het model overeenkomen met de werkelijke waarden.
4.1 Interpretatie van de R²-waarde
- R² = 0: Het model verklaart geen variabiliteit in de afhankelijke variabele. De voorspellingen zijn niet effectief.
- R² = 1: Het model verklaart 100% van de variabiliteit in de afhankelijke variabele. De voorspellingen komen perfect overeen met de werkelijke waarden.
- 0 < R² < 1: Het model verklaart een deel van de variabiliteit. Hoe dichter de R²-waarde bij 1 ligt, hoe beter het model presteert.
De R²-waarde geeft ons dus een duidelijk beeld van hoe accuraat en betrouwbaar de voorspellingen van ons model zijn. Hoe hoger de R²-waarde, hoe meer vertrouwen we kunnen hebben in de resultaten van het model.
Meer info? hier
4.2 Validatiestappen
- Maak een FeatureCollection die zowel de voorspelde als de oorspronkelijke (GEDI)
rh98waarden bevat. Dit kan op 2 manieren (zie ook Classificatie):
// Voeg de 'voorspelde' waarden samen met de 'validatiewaarden' via SampleRegions
var predictedValidation = regression.sampleRegions({collection: validation, geometries: true});
Dit is hetzelfde als:
// Dit is hetzelfde als (zoals in de classificatie):
var validation_S2 = Sentinel_2021.sampleRegions({
collection: validation,
properties: ['rh98'], //'Waarde uit de validatie GEDI-shot'
scale: 25, // 25m = radius GEDI-shot
tileScale: 16
})
var predictedValidation = validation_S2.classify(RF_regressor, 'predicted') // 'predicted' hernoemt de band
- Bereken de R²
De code voor het berekenen van de R²-score is hieronder gegeven. Dit maakt gebruik van de .reduceColumns()functie, de pearson-correlatie tussen 2 eigenschappen kan berekenen. Hier= predicted en rh98.
// Berekening van R²
var pearsonCorrelation = predictedValidation.reduceColumns({
reducer: ee.Reducer.pearsonsCorrelation(),
selectors: ['predicted', 'rh98']
}).get('correlation'); // Pearson's correlation coefficient
// R2 is het kwadraat van de Pearson correlatie
var R2 = ee.Number(pearsonCorrelation).pow(2);
// Print de R²-waarde naar de console
print('R²-waarde:', R2)
Grafiek; voorspelde vs GEDI rh98
// Maak een grafiek aan die de voorspelde versus validatiewaarde bevat;
var chartTraining = ui.Chart.feature.byFeature(predictedValidation, 'predicted', 'rh98')
.setChartType('ScatterChart').setOptions({
title: 'Predicted vs Observed - Training data ',
hAxis: {title: 'observed', viewWindow: {min: 0, max: 50},},
vAxis: {title: 'predicted', viewWindow: {min: 0, max: 50},},
pointSize: 3,
trendlines: { 0: {showR2: true, visibleInLegend: true} ,
1: {showR2: true, visibleInLegend: true}}});
print(chartTraining);
EXTRA - Stap 9: Identificatie van de meest belangrijke banden
// Met de .Explain()-functie kunnen we informatie uit de Random Forest regressor krijgen
var dict = RF_regressor.explain();
print("Classifier information:", dict);
var variableImportance = ee.Feature(null, ee.Dictionary(dict).get('importance'));
// Maak een grafiek aan die meest belangrijke banden bevat
var chart = ui.Chart.feature.byProperty(variableImportance)
.setChartType('ColumnChart')
.setOptions({
title: 'Random Forest Variable Importance',
legend: {position: 'none'},
hAxis: {title: 'Bands'},
vAxis: {title: 'Importance'}
});
print(chart);
AGBD - kartering
Tracht nu ook een 'above ground biomass density' (agbd) kartering te maken, door gebruik te maken van de GEDI-L4A dataset.
Antwoord: https://code.earthengine.google.com/8e9858901bb1b025d781af7c79b60114